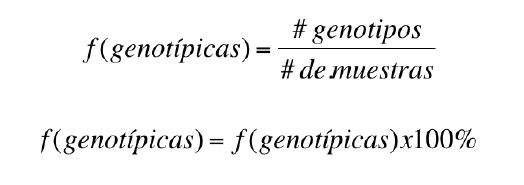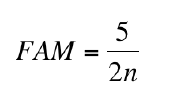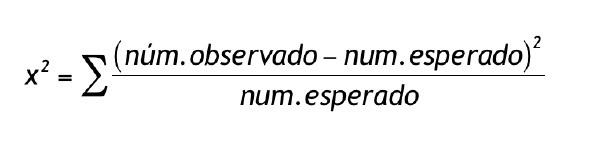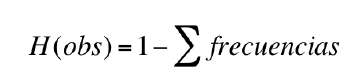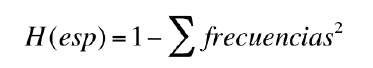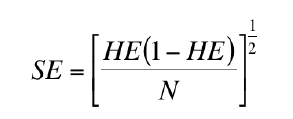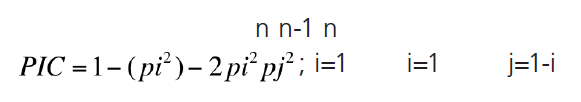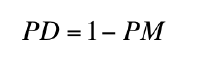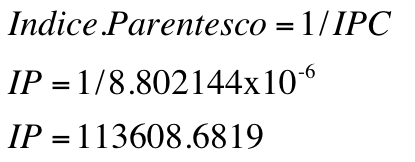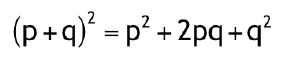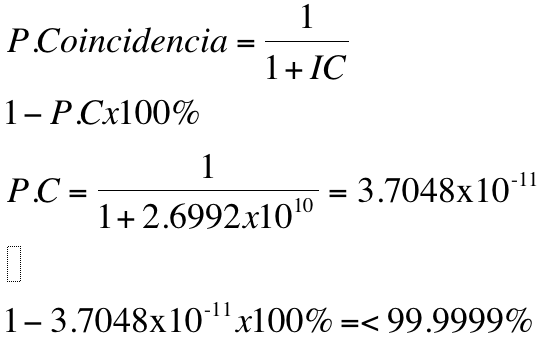Keywords
DNA markers, forensic genetic, PCR, allelic frequency, genotypes, database.
Introducción
Genética
La genética es el estudio de la herencia y la variación biológica en los organismos, puede ser considerado como una base individual, donde los rasgos particulares son heredados de padres a hijos o más global como un rastreo del movimiento de marcadores genéticos en una población. Todos los organismos se componen de células (la unidad más pequeña de la vida), en las células humanas el ADN localizado en el núcleo es denominado ADN nuclear organizado en cromosomas [13, 17]. El genoma humano consta de 22 pares de cromosomas autosómicos y dos cromosomas sexuales, en hombres es designado como XY, mientras que en mujeres es XX. Muchas pruebas de identificación humana son realizadas usando marcadores en cromosomas autosómicos y determinando el género sobre los cromosomas sexuales [3-6]. El ADN se compone por regiones “codificantes” (conocidas como genes) y “no codificantes” (ADN basura). Los marcadores usados en pruebas de identificación humana se encuentran en las regiones no codificantes o dentro de los genes que no codifican en las variaciones genéticas. Las pequeñas variaciones en el ADN individual permite la diferencia entre las personas, las diferentes formas de un mismo gen o marcador es llamado alelo, si los alelos de un lugar en particular (locus) en el genoma son los mismos en ambos pares de cromosomas, la situación es llamada homocigoto, si una pequeña diferencia está presente en un locus en específico, la diferencia alélica será llamada heterocigoto [13, 11].
Identificación Humana
El estudio de la variabilidad genética como medio de identificación humana inició con el análisis de los grupos sanguíneos, continuando con proteínas séricas leucocitarias y eritrocitarias más tarde el sistema de Antígenos Leucocitarios Humanos (HLA). En 1985 Alec Jeffreys y colaboradores describían un método de identificación al que denominaron DNA finger printing o huella genética; que sería la solución definitiva al análisis de la diversidad humana en Medicina Legal, Investigación Biológica de Paternidad y en criminalística. En 1987, el uso de la “huella genética” se admitió en procesos penales en Inglaterra, Estados Unidos de América y actualmente la prueba de ADN está consolidada científicamente y de gran peso ante los tribunales a nivel mundial. El avance tecnológico y científico en marcadores genético moleculares han permitido el desarrollo de la Genética Forense, para la identificación de personas, restos cadavéricos, análisis de vestigios biológicos de interés criminal, investigación de paternidad y las variabilidades genéticas de la población [10].
Genotipo y Fenotipo
El genotipo es el contenido genético de un individuo en forma de ADN y se define como el conjunto de genes de un organismo [9], el fenotipo es la manifestación física o externa de un gen.
Genética poblacional
La genética de poblaciones no solo estudia la composición genética del individuo, también a la población entera y los cambios de esta a través de las generaciones, y establecer la variación de distintos locus, tratar de explicar las posibles causas de esa variación e incluso establecer un modelo, siendo aplicable a todos los seres vivos [12]. Un alelo se define como las múltiples variaciones de un gen o secuencia de ADN en la población y cada par de alelos conformará un genotipo (combinación de alelos para un gen o grupo de genes). Todos los marcadores genéticos se encuentran en la población con determinada frecuencia, siendo importante determinar su presencia. Existen cuatro fuerzas a considerar: la selección, deriva génica, mutación y migración. La frecuencia de un genotipo (homocigoto o heterocigoto), varía al cambiar las frecuencias alélicas. Si existen más de dos alelos de un mismo gen, éste es considerado como polimórfico y cada nuevo alelo que surge en la población aumenta el polimorfismo del gen, para considerarlo como tal, la frecuencia del alelo más común debe ser menor al 99%, y para un alelo poco común debe superar al menos el 0.005% de frecuencia en la población; los alelos que no alcanzan estas frecuencias son considerados como raros [12].
ADN microsatélite (STR). Por su tamaño son conocidos como Repeticiones Cortas en Tándem, son elementos extraordinariamente útiles en la identificación humana o en el mapeo genómico, debido al elevado polimorfismo, con tasas de mutaciones relativamente bajas y son de tamaño pequeño lo que optimiza su amplificación y ubicación cromosómica establecida. Formados por secuencias muy cortas de ADN repetitivo (2 a 13 pb), distribuidos ampliamente en el genoma en bloques menores a 400 pb, con tamaños de 80 y 550 pb. Los STRs son la principal herramienta de la Genética Forense por sus características y también que es posible tipificar muestras con poco material biológico por sistemas de PCR múltiplex.
Ventajas de los Marcadores STR
Son marcadores populares para tipificar ADN forense, trabaja con poca cantidad de templados de ADN o muestras con ADN degradado, son manejables para la automatización e involucra detección sensible de fluorescencia, permitiendo recolectar datos más rápidamente, son altamente discriminatorios entre individuos no relacionados y más estrechos entre individuos relacionados. Los alelos hacen que los resultados sean más fáciles de interpretar y de compararlos al usar una base de datos de ADN computarizados [3-6].
Los 13 str loci del codis
El proyecto para la creación de una Base de Datos a nivel Nacional en Estados Unidos involucrando los STR, comenzó en abril de 1996 y concluyo en noviembre de 1999 participando 22 laboratorios de tipificación de ADN y la evaluando 17 candidatos de STR loci. Los STR evaluados fueron: CSF1PO, F13A01, F13B, FES/FPS, FGA, LPL, TH01, TPOX, vWA, D3S1358, D5S818, D7S820, D8S1179, D13S317, D16S539, D18S51 y D21S11 (Butler, 2005). Los 13 STR loci elegidos para formar parte de la base de datos nacional de ADN del CODIS (Combined DNA Index System) fueron: CSF1PO, FGA, TH01, TPOX, VWA, D3S1358, D5S818, D7S820, D8S1179, D13S317, D16S539, D18S51 y D21S11. Los 3 marcadores con mayor polimorfismo son: FGA, D18S51 y D21S11, TPOX muestra la menor variación entre individuos [3, 4, 5, 65)16] En la siguiente imagen se muestran las posiciones cromosómicas de los loci (Figura 1).
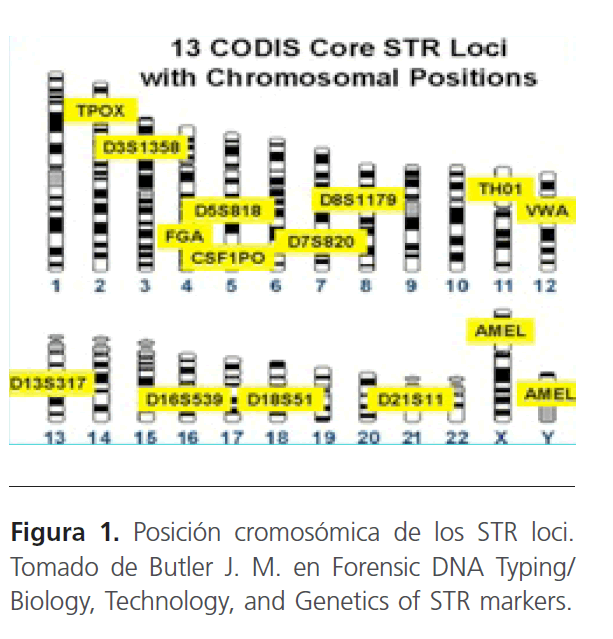
Figura 1: Posición cromosómica de los STR loci. Tomado de Butler J. M. en Forensic DNA Typing/ Biology, Technology, and Genetics of STR markers.
Objetivo del presente estudio
El objetivo del presente estudio es obtener una base de datos con una muestra representativa de la población Zacatecana mediante la recopilación de muestras de los 58 municipios que componen el estado y obtener el perfil genético de cada muestra. Obtener las frecuencias alélicas y genotípicas para crear una base de datos que pueda ser utilizada para cálculos estadísticos en casos forenses, empleando las frecuencias obtenidas, los resultados serán más exactos para establecer las probabilidades de parentesco y/o paternidad, poder de discriminación e índice de coincidencia de los diversos casos forenses.
Metodología
Obtención de muestras: El número de muestras a estudiar fue un total de 271 muestras de las que 170 pertenecen a hombres (62.73% de las muestras) y 101 a mujeres (37.27% de las muestras)e este número que fue obtenido con la información obtenida con el censo realizado por el INEGI en el 2005, con una población de1,37,692 habitantes, estableciendo un 5% de error y un 95% de confianza. Las muestras son aceptadas tomando en cuenta todos los requisitos planteados anteriormente y abarcando todos los municipios del estado de Zacatecas. Las muestras se colectaron mediante hisopados bucales y sangre en tarjetas FTA™, asignando una clave a cada muestra recabada.
Extracción con Chelex®100
Introducida en 1991 a la comunidad de ADN forense, Chelex®100 (Bio-Rad Laboratorios, Hércules, CA), es una resina con un cambio de ión que es agregada en suspensión a las muestras. El Chelex está compuesto de copolímeros de estireno divinilbenceno contiene iones apareados de iminodiacetato que actúan como grupos quelantes en la unión de iones metálicos polivalentes como el Mg (magnesio), estos iones de Mg son sobre dibujados y colocados por encima del ADN, y se remueven mediante enzimas del ADN como las nucleasas que se activan y las moléculas de ADN quedan protegidas [5)] Las resinas quelantes en suspensión pueden ser agregadas directamente a muestras biológicas como: sangre líquida, sangre en soporte, o semen. Las muestras biológicas como la sangre en soportes son tratadas con Chelex al 5% y colocadas a ebullición por varios minutos para romper las células y liberar el ADN, posteriormente se realizan lavados para remover contaminantes e inhibidores (grupo hemo y otras proteínas). Posteriormente, se centrífuga, la resina junto con restos celulares quedan sedimentados en el fondo del tubo, el sobrenadante contiene el ADN que debe ser removido y puede agregarse directamente a la reacción de PC5)[14].
Papel FTA
Mediante una lanceta se punza el dedo donador colocando la sangre sobre el papel FTA™ al contacto éste lisa las membranas celulares de la sangre y desnaturaliza las proteínas, los ácidos nucleicos son inmovilizados, protegidos de los rayos UV, agentes microbianos y hongos [14, 5)]
Amplificación de ADN con marcadores STR’s por PR
Un método eficaz para amplificar segmentos específicos de ADN: la Reacción en Cadena de la Polimerasa ó PCR (por sus siglas en inglés Polymerase Chain Reaction), fue descrita por primera vez en 1985 por Kary Mullis (premio nobel en Química en 1993) y miembros del grupo de Genéticos Humanos de la Corporación Cets, [185)3, 4, 5, 6] En muestras de ADN forense debido a la alta sensibilidad y rapidez, no está limitada por la calidad del ADN [3-5 7,0)].
Kit para la Amplificación de ADN
El kit AmpFlSTR Identifiler® presenta un poder de discriminación de 7.9x10-19, con 15 marcadores STR y uno más para Amelogenina incluyendo los 13 marcadores STRs para la creación de la base de datos del CODIS (D8S1179, D21S11, D7S820, CSF1P0, D3S1358, TH01, D13S317, D16S539, D2S1338, D19S433, vWA, TPOX, D18S51, D5S818, FGA y Amelogenina) (Marcador De Posición 2)m;2z,15)
Loci Genéticos Deseados
Los 13 core STR loci siguientes han sido elegidos para ser la base de la futura base de datos na cional de ADN del CODIS: CSF1PO, FGA, TH01, TPOX, vWA, D3S1358, D5S818, D7S820, D8S1179, D13S317, D16S539, D18S51 y D21S11 [14, 15) (estos son empleados en estudios para crear bases de datos), y otros tres marcadores más: D2S1338 Y D19S433, y uno extra para Amelogenina dan un total de 16 marcadores analizados.
Tipificación de ADN mediante el Secuenciador Automatizado 3130 de Applied Biosystems®.
El uso del secuenciador automatizado y los software Genotyper® y GeneMapper® asignan el valor alélico correspondiente a cada una de las muestras para los diferentes marcadores analizados.
Cálculos Estadísticos
El equilibrio Hardy Weinberg (H-W): los alelos de un locus muestran correlación no a priori con cada otro locus [2)]. El equilibrio de Hardy Weinberg ocurre en una población que se mezcla al azar y no ocurren las fuerzas de migración, deriva génica o selección en una población infinita; el resultado es un locus en equilibrio donde las frecuencias alélicas no cambian de generación en generación, las frecuencias genotípicas se mantienen constantes, la suma de las frecuencias genotípicas de un locus y de los alelos siempre debe ser uno [5)].
Estimación de Frecuencias
La frecuencia génica ó alélica es la medida de la proporción relativa de alelos de una población dada, expresándose en porcentaje o en la unidad. Se estima contando el número de veces que es observado el alelo de un locus y dividiéndolo entre el número total de alelos estudiados [4, 5, 6, 16).

Las frecuencias genotípicas se obtienen mediante a partir de dividir cada número de combinaciones observadas (genotipos) entre el número total de las muestras analizadas y posteriormente multiplicando por 100% para obtener el porcentaje de las frecuencia genotípicas.
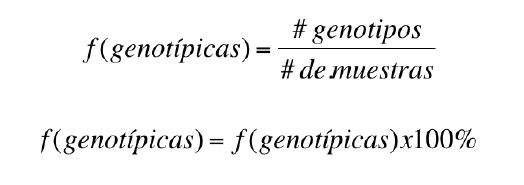
Algunos alelos ocurren (aparecen) en menor frecuencia que otros por lo que la frecuencia mínima para estos alelos se obtiene de dividir cinco entre 2n donde “n” es el numero de muestras totales analizadas para la base de datos.
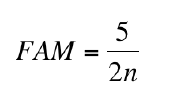
* La frecuencia alélica mínima para aquellos alelos que no aparecen regularmente es 9.2251x10-3 ó 0.0092.
Prueba del Equilibrio de un Locus. No hay forma de comprobar los supuestos de Hardy-Weinberg contra una muestra de datos observados a menos que los fenotipos dominantes hayan sido analizados mediante observaciones. Al involucrar los alelos codominantes se puede confirmar fácilmente las observaciones contra los valores esperados en equilibrio a través de la prueba de Chi-cuadrada [5)].
La Prueba de Chi-Cuadrado. En cualquier experimento genético es necesario decidir si nuestros datos están en relación con las proporciones Mendelianas. Una prueba estadística que resulta muy útil es la prueba de hipótesis de Chi-cuadrad8) [19].
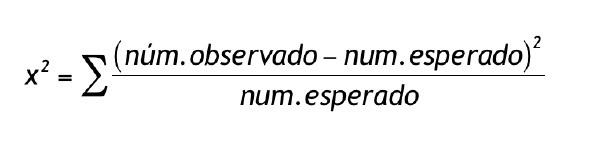
Grados de Libertad. El número de variables en las pruebas de Chi-cuadrada en el equilibrio de Hardy- Weinberg consiste en el número de fenotipos menos 1. El número de variables observadas (número de fenotipos =k) se restringe comprobando su conformidad con las proporciones de frecuencias esperadas Hardy-Weinberg originadas por el numero de variables adicionales (número de alelos o frecuencias de alelos =r). Se tienen (k-1) grados de libertad en el número de fenotipos, (r-1) grados de libertad al establecer las frecuencias de los alelos r. El número combinado de grados de libertad es (k-1) – (r-1) = k-r. En la mayoría de las pruebas de Chi-cuadrada para equilibrio que involucran alelos múltiples, el número de grados de libertad es el número de fenotipos menos el número de alelos [15).
Grados de Libertad= n-1 n= es el número de clases
La Probabilidad de Coincidencia (PM) es la probabilidad de que dos personas tomadas al azar posean el mismo genotipo descrito para la población de estudio.
Heterocigosidad (H) representa una mejor medida de la variación genética, ya que es precisa y no arbitraria. Estudiada como HO y HE, se define como HO la frecuencia relativa de individuos heterocigo- tos observados en la muestra para cualquiera de los loci y se obtiene de:
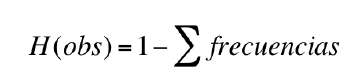
Heterocigosidad Esperada (HE), es la probabilidad de que dos alelos tomados al azar de la población sean diferentes obteniéndose a partir de la siguiente fórmula:
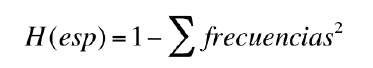
El Error Estándar o SE es para estimar el error de HE y se obtiene a partir de:
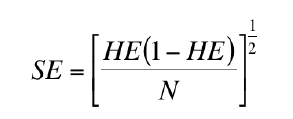
Donde; N es el número de muestras estudiadas.
Realizando este cálculo de manera manual para cada marcador analizado con sus respectivos datos.
Índice de Contenido Polimórfico (PIC) es similar al valor de heterocigosidad y oscila entre 0 y 1. Este índice evalúa la información de un marcador en la población de acuerdo a las frecuencias de los alelos. Se obtiene de multiplicar la probabilidad de cada posible cruzamiento (estimado a partir de las frecuencias alélicas) por la probabilidad de que sean informativos, es decir, que se pueda identificar al progenitor del que procede el alelo.
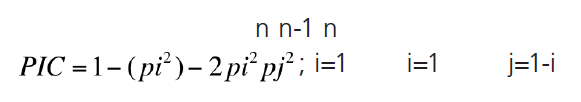
Donde pi...pn son las frecuencias de los n alelos.
Poder de Discriminación (PD) es la probabilidad de que dos individuos no relacionados y tomados al azar puedan ser diferenciados genéticamente mediante el análisis de un marcador o marcadores, calculándose:
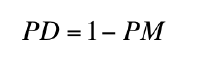
Resultados
Una vez que las muestras fueron recabadas para realizar el estudio genético poblacional, se realizó la extracción del ADN de las muestras para ser amplificadas, genotipificadas y posteriormente obtener los perfiles genéticos de las 271 muestras colectadas, se analizó cada uno de los alelos observados para cada locus concentrando los datos para posteriormente obtener las frecuencias alélicas de cada uno de ellos. Estos datos se encuentran agrupados en la Tabla 2 y Tabla 3 (que se encuentran como anexos al final del escrito).
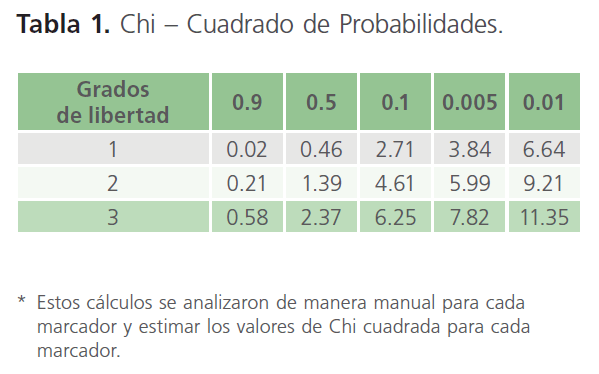
Tabla 1: Chi – Cuadrado de Probabilidades.
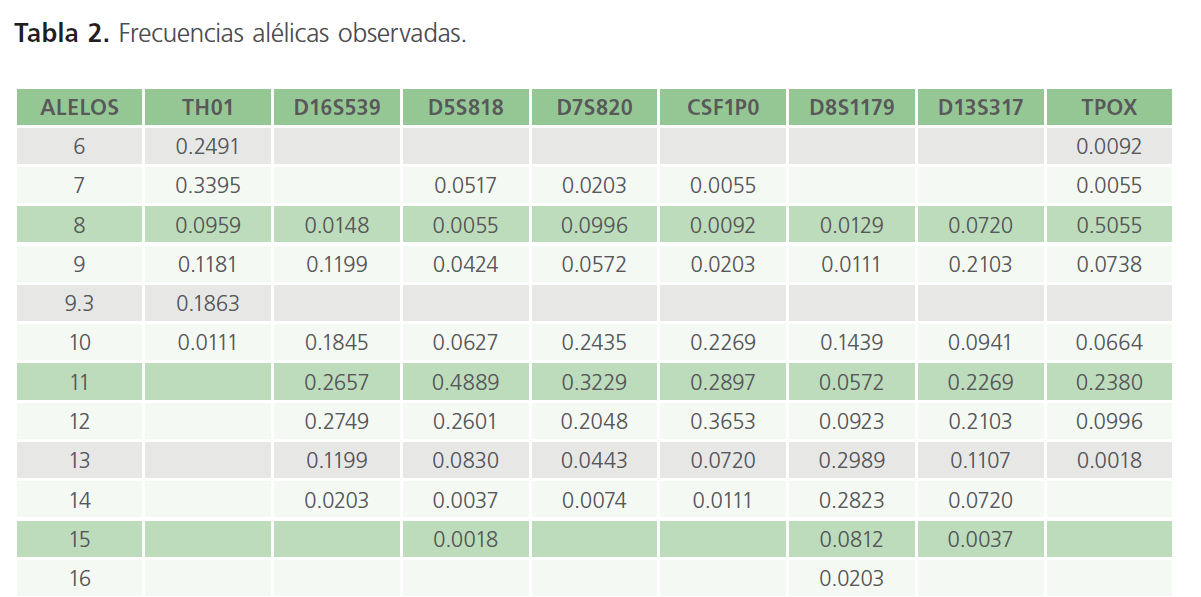
Tabla 2: Frecuencias alélicas observadas.
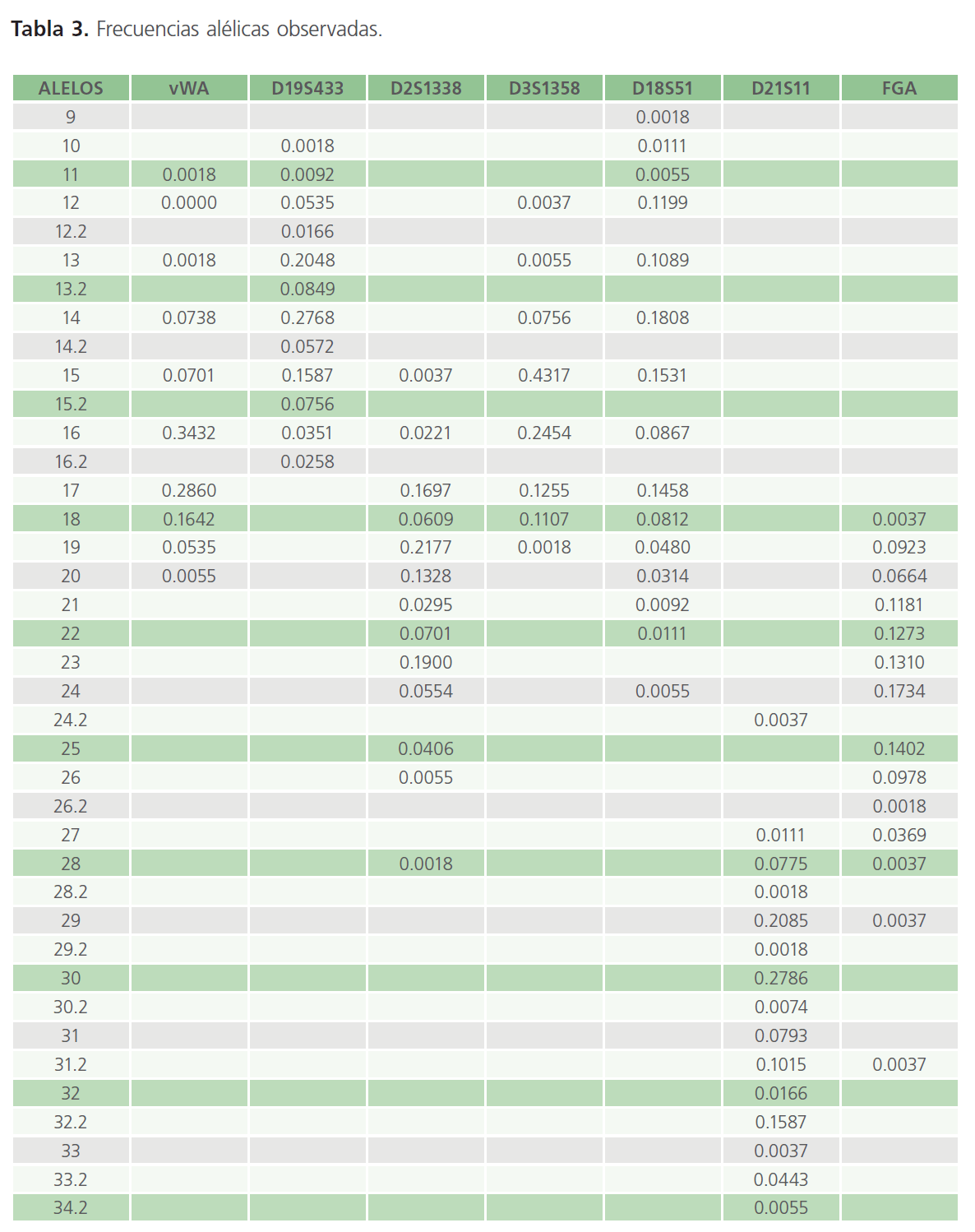
Tabla 3: Frecuencias alélicas observadas.
Una vez realizado el estudio del equilibrio se obtuvieron diversos parámetros estadísticos de interés que nos permiten saber cómo se están comportando los diferentes alelos dentro de la población concentrados en Tabla 4 y Tabla 5 (anexo).
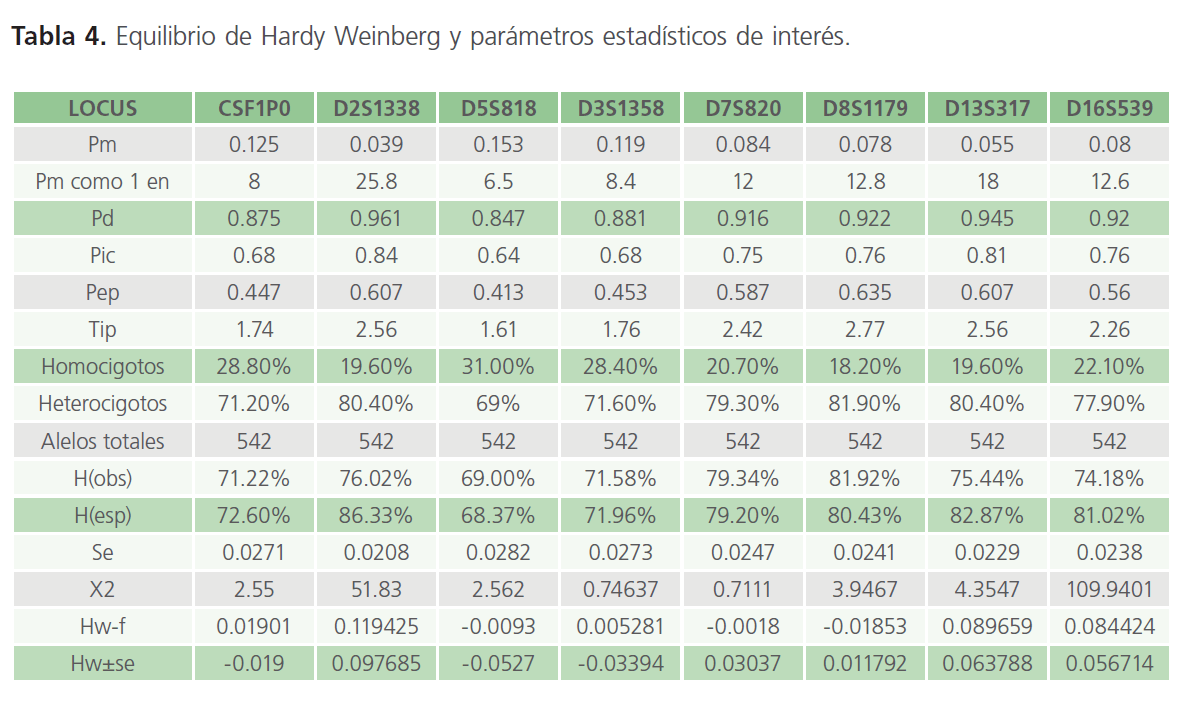
Tabla 4: Equilibrio de Hardy Weinberg y parámetros estadísticos de interés.
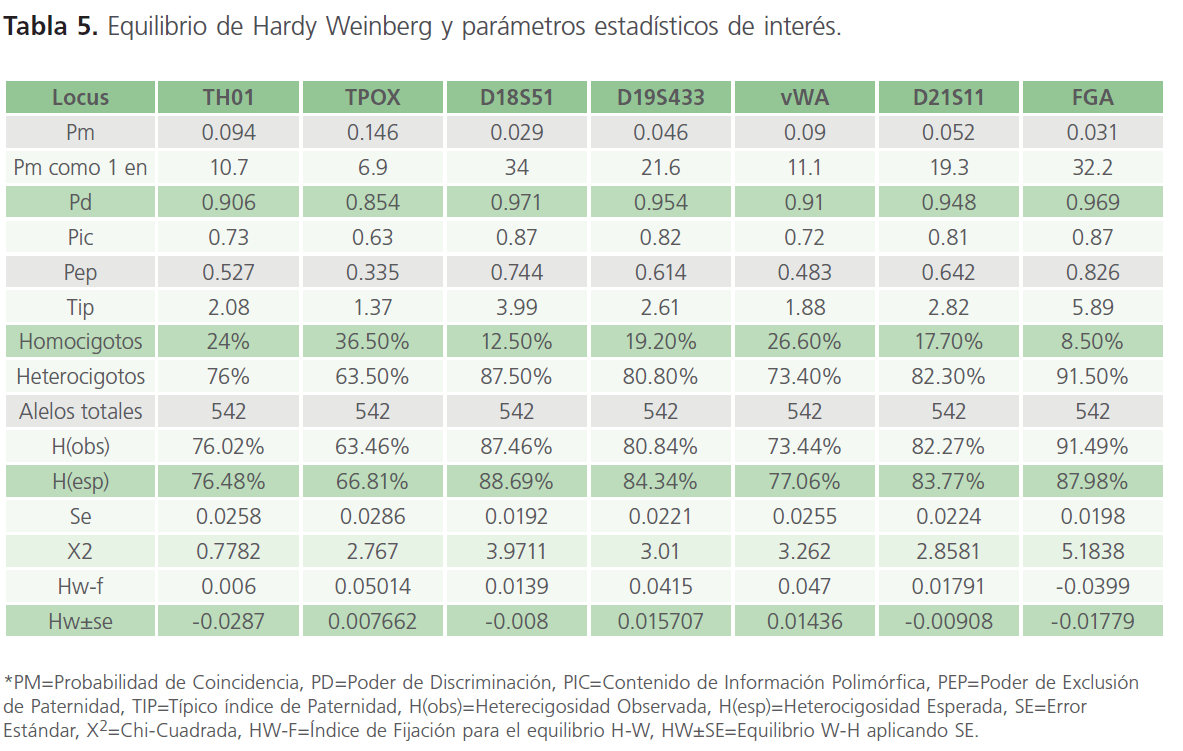
Tabla 5: Equilibrio de Hardy Weinberg y parámetros estadísticos de interés.
Una vez obtenidas las frecuencias alélicas encontradas dentro de la población Zacatecana se realiza una comparación con las frecuencias proporcionadas por el manual de Identifiler® y las encontradas en el estudio de la Población Mestiza del Noroeste del País, dichos resultados se encuentran concentrados en la Tabla 6; (anexo).
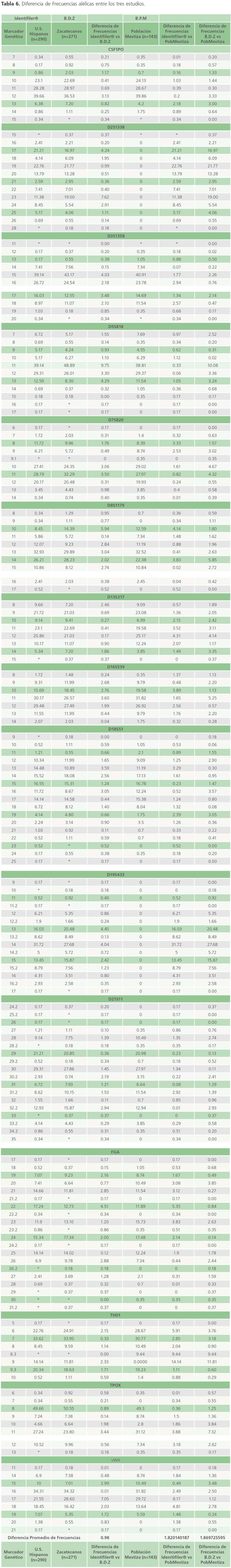
Tabla 6: Chi – Cuadrado de Probabilidades.
Discusión
Actualmente son pocos los estudios realizados sobre la población Mexicana que proporcionen datos estadísticos sobre las frecuencias alélicas y génicas de la población, ya que son pocos los laboratorios con equipos y recursos necesarios para realizar este tipo de estudios y el elevado costo para llevarlos a cabo. Pese a eso diversas Procuradurías como: Procuraduría General de la República, Procuraduría General de Justicia Militar, Procuraduría General de Justicia del Distrito Federal y las Procuradurías Generales de Justicia de los treinta y un Estados integrantes de la Federación, celebraron un Convenio de Colaboración llevado a cabo en la ciudad de San Luis Potosí, S. L. P., el veintisiete de Marzo del año 2007, publicado en el Diario Oficial de la Federación con fecha Veintiséis de junio del mismo año; firmando de común acuerdo tanto Directores de los Servicios Periciales como Procuradores de la Federación, en donde quedó establecido el material para la recolección de las muestras y para la determinación de los perfiles genéticos, que permitirán establecer la futura base de datos a Nivel Nacional de la Población Mexicana. Con ello se realizó un Anteproyecto de Ley para la Creación de la Base Nacional de Datos Genéticos Forenses en la que se exponen las ventajas de contar con una Base de Datos a Nivel Nacional para el esclarecimiento de hechos delictuosos. Para ello se decidió utilizar un procedimiento de extracción, amplificación y tipificación de ADN que fuera eficiente y que brindara resultados óptimos debido a la gran cantidad de muestras que se decidieron analizar. El análisis realizado para 15 STRs contenidos en el kit AmpFlSTR® Identifiler® de la compañía de Applied Biossystems® permitió realizar la tipificación para 271 muestras de la población del Estado de Zacatecas realizándolo de manera automatizada, lo que permite disminuir el error por manejo y apreciación de los resultados; utilizando este kit por contener todos los loci deseados y seleccionados para la creación de la base de datos del FBI llamada CODIS y tres más (incluyendo uno para Amelogenina). Se realizó la tipificación automatizada mediante el secuenciador ABI 3130 HITACHI de la compañía de APPLIED BIOSYSTEMS ® se obtuvo el genotipo para cada muestra, posteriormente se analizaron y organizaron todos los genotipos de las muestras, para establecer las frecuencias alélicas y genotípicas dentro de la población Zacatecana. Comparando las frecuencias alélicas obtenidas de este estudio y las reportadas en el Manual de Identifiler® (realizado con una muestra de 290 Hispanos radicados en Estados Unidos), a la par se compararon las frecuencias reportadas en un estudio de la población mestiza del noroeste del país y las obtenidas del manual de Identifiler®, realizando de igual manera una comparación entre las frecuencias obtenidas de la población Zacatecana y las reportadas en el estudio de la población mestiza encontrando resultados interesantes que se encuentran reportados en conjunto en la Tabla 6. Al analizar esta tabla de comparaciones se observa para los diferentes marcadores analizados que en:
• CSF1P0 el alelo 15 solo se encontró en el estudio de los hispanos.
• D2S13338 este marcador no fue analizado para la población mestiza, pero si para los hispanos y los zacatecanos (reportando los alelos 15 y 28 para esta última).
• D3S1358 en la base de hispanos el alelo 20 está reportado sin detectarlo en la población zacatecana.
• D5S818 para la población zacatecana y mestiza los alelos 16 y 17 no se presentaron, pero en el grupo de hispanos analizados si se reportan.
• D7S820 en los hispanos los alelos 9.1 y 15 no se reportan, coincidiendo con la población zacatecana añadiéndose el alelo 6, en la población mestiza los alelos ausentes son 6 y 15.
• D8S1179 en los hispanos los alelos 18 y 19 no se detectaron, en la población zacatecana no se detectaron los alelos 17, 18 y 19, la población mestiza coincidió con la ausencia de estos alelos sumándose el alelo 9.
• D13S317 tanto en hispanos como en mestizos no se encontró el alelo 15 pero si se en la población zacatecana.
• D16S539 las tres poblaciones estudiadas no presentaron los alelos 5 y 15.
• D18S51 tanto en hispanos y mestizos no se encontraron los alelos: 7, 9, 10.2, 13.2 y 14.2 sumándose el alelo 23 y 25 para la población mestiza, y en los zacatecanos no se encontraron los alelos 7, 10.2, 13.2, 14.2, 23 y 25.
• D19S433 en mestizos este marcador no fue analizado, en hispanos y zacatecanos no presentaron los alelos 17.2 y 18.2, el alelo 10 tampoco se encontró en hispanos, los alelos 9, 11.2, y 17 se sumaron a los alelos ausentes para los zacatecanos.
• D21S11 los alelos ausentes que coinciden en los tres estudios son: 24, 24.3, 25, 29.3, 34, 34.1, 35.1, 35.2, 36, 37 y 38. Para la población de Zacatecas y población mestiza los alelos faltantes son: 25.2, 26 y 35 el estudio en los hispanos no se encontró el alelo 28.2 mientras que para las otras poblaciones si se detectó. En la población mestiza el alelo 33 no estuvo presente dentro del estudio mientras que en la población zacatecana e hispana si se detectó.
• FGA tanto para los hispanos, zacatecanos y la población mestiza comparten la ausencia de los alelos: 16, 17.2, 18.2, 19.2, 20.2, 22.3 y 43.2. Entre la población zacatecana y la población mestiza comparten por ausencia los alelos: 21.2, 22.2, 24.2 y 30.2. Los hispanos y la población mestiza no presentaron el alelo 29, la población zacatecana e hispanos no presentan el alelo 30. En el estudio de los hispanos no hay dato para el alelo16.1, para la población zacatecana no se encontraron los alelos 23.2 y 32.2.
• TH01 los alelos que comparten los tres estudios por la ausencia de éstos son: 4, 11 y 13.3, el alelo que no está presente en hispanos y zacatecanos es el 8.3, los zacatecanos y mestizos no presentaron el alelo 5.
• TPOX en hispanos el alelo 13 no está presente y en la mestiza el alelo 7.
• vWA en los 3 estudios no se presentaron los alelos: 12, 13, 22, 23, y 24, el alelo 21 no se encontró en la población zacatecana y mestiza, el alelo 11 y 20 no se reportan en la población mestiza mientras que en hispanos y zacatecanos si se detectó.
Realizando una comparación entre las frecuencias obtenidas del manual de Identifiler® y las obtenidas mediante el análisis de la población Zacatecana el promedio de la diferencia de las frecuencias es de 0.98% (en una escala del 1 al 100, siendo mínima), al comparar las frecuencias de la población mestiza con las obtenidas del manual de Identifiler® el promedio de la diferencia de las frecuencias es de 1.82%, y finalmente al comparar las frecuencias obtenidas de la población Zacatecana y las de la población mestiza el promedio de las diferencias de las frecuencias es de 1.87%. La diferencia entre las frecuencias encontradas es mínima, existe mayor diferencia entre los datos obtenidos de la población mestiza y la población zacatecana, la diferencia de las frecuencias entre la población zacatecana y los datos obtenidos de los hispanos es 50% menor que los datos de la población mestiza y la hispana. Los datos encontrados en este estudio se asemejan más a los obtenidos de los hispanos (reportados en el manual de Identifiler®), encontrando algunos de los alelos entre la población zacatecana que no se reportaron entre los hispanos.
La Probabilidad de Coincidencia obtenida mediante este análisis es significativamente mayor que el registrado por el análisis de las muestras de los hispanos. Mientras que para la población Zacatecana es de 1/6.7965-18, proporcionando una probabilidad de coincidencia de 1.471217 (superando ampliamente la población a nivel mundial que es de 7,000 millones de acuerdo con datos de la ONU) para los datos obtenidos de los hispanos la probabilidad de coincidencia es de 1/1.31x1017, haciendo una diferencia de 0.1612x1017, entre los datos obtenidos de la población Zacatecana y de los hispanos.
Si bien no existen otros datos para cotejar los resultados obtenidos para Chi-Cuadrada y poder observar las fluctuaciones de los datos obtenidos mediante este análisis lo que si podemos recalcar es que las frecuencias obtenidas mediante la plantilla de Excel® de Microsoft Word® y las obtenidas de manera manual son casi idénticas, los datos que se analizaron manualmente se opto por dejar cuatro dígitos y no presentarlas en forma de porcentaje en cambio, las obtenidas mediante la plantilla de Excel® son presentadas en forma de porcentaje y redondeando las cifras obtenidos a dos dígitos. El uso de la plantilla de Excel® proporcionada por la página de Internet de la compañía de Promega® (antes citada) ayudó a proporcionar un análisis más rápido debido a la gran cantidad de genotipos por analizar de las diferentes muestras ayudando así a disminuir el tiempo de análisis de éstas y a obtener los parámetros estadísticos que facilitan el análisis de muestras forenses como los son: Probabilidad de Coincidencia, Poder de Discriminación, Contenido de Índice Polimórfico, Poder de Exclusión de Paternidad y Típico Índice de Paternidad, (Tabla 4 y Tabla 5). Estos parámetros deben ser obtenidos ya que en casos de probabilidad de coincidencia nos permiten saber que tan probable es que dos personas tomadas al azar pudieran tener el mismo genotipo. El poder de exclusión es el promedio de todas las posibles combinaciones de madre e hijo para la identificación de un padre alegado que será excluido de la prueba de paternidad antes de ser sometido al análisis de ADN. El índice de paternidad nos indica la probabilidad que tiene el presunto padre de ser el padre biológico del hijo con respecto a una hombre tomado al azar. El contenido de índice polimórfico es similar a los cálculos de heterocigosidad proporcionando la información para cada marcador en la población de acuerdo a las frecuencias de los alelos. Los datos obtenidos para Homocigotos y Heterocigotos proporcionan el porcentaje de éstos que están presentes en el tamaño de muestra analizada aprovechándose para observar si la población se encuentra inclinada hacia una mayor cantidad de homocigotos o heterocigotos.
La cantidad de Heterocigotos Observada, Heterocigotos Esperados y el Error Estándar son datos que son necesarios obtener y analizar si se quiere evaluar el Equilibrio de Hardy–Weinberg. Una vez que se analizaron las frecuencias de la población Zacatecana también se observa si la población de Zacatecas se encuentra en equilibrio de H-W, cuyo único propósito de este análisis era establecer una Base de Datos para la población de Zacatecas que pudiera ser empleada en el análisis Forense.
En la práctica forense
Caso1. Vínculo biológico
Problema: Se presenta un caso donde el Problema de Estudio es Establecer si existe Vínculo Biológico entre dos personas del sexo Femenino(X,X) que presumían tener parentesco con uno de los cadáveres procedentes de un accidente tipo choque entre dos vehículos cuya identificación no podía realizarse mediante el reconocimiento de los cuerpos por los familiares debido a que después del suceso los vehículos se incendiaron resultando difícil su reconocimiento optando entonces realizar un estudio genético entre los tres cadáveres obtenidos del accidente.
Material de estudio: Las muestras de referencia (sangre en papel FTA®, cabello e hisopados bucales) son recibidas junto con tres muestras de tejido biológico pertenecientes a los cadáveres identificados como cadáver 1(uno), 2(dos) y 3(tres).
Análisis estadístico: Para realizar los cálculos estadísticos de vínculo biológico se empleó la Tabla 6 de frecuencias alélicas obtenida mediante el estudio de la Población Zacatecana. Los perfiles obtenidos de las muestras analizadas se reportan en la Tabla 7 (anexa al final de la bibliografía). De manera simultánea se analizaron controles (+) y (-).
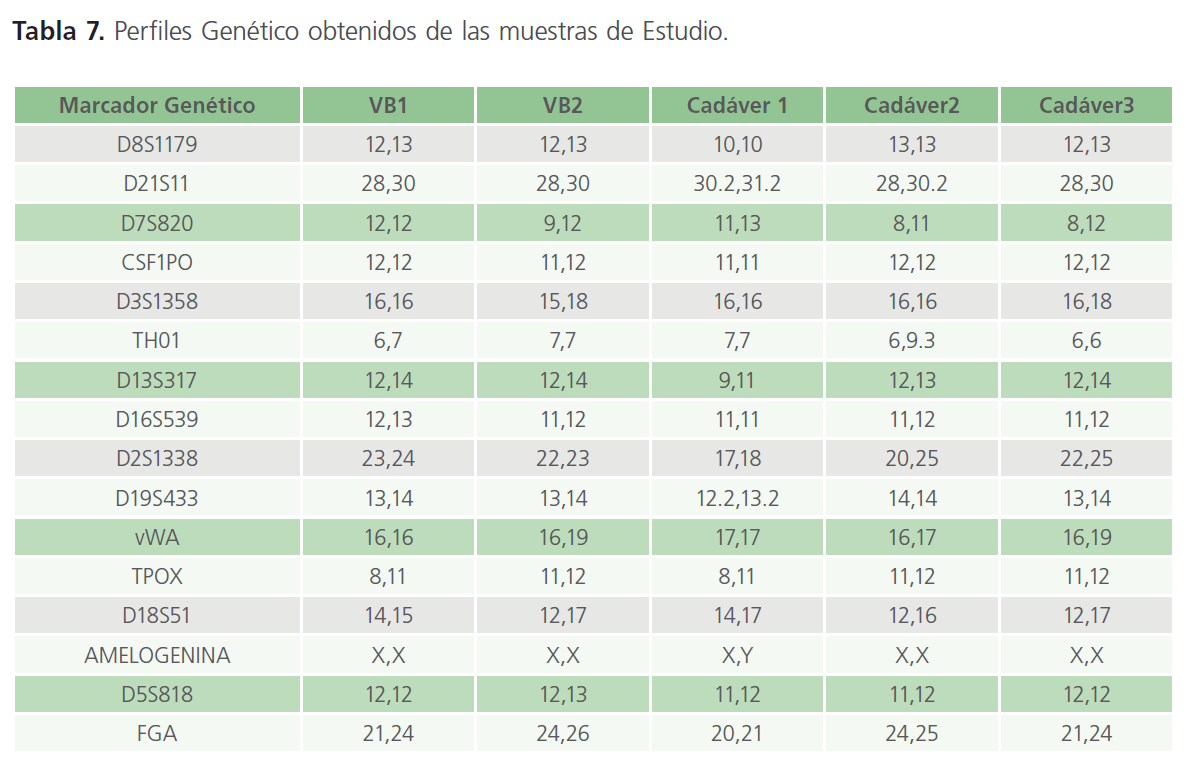
Tabla 7: Perfiles Genético obtenidos de las muestras de Estudio.
Posteriormente, se establece el índice de parentesco de acuerdo al genotipo presentado en las muestras. Resultados e interpretación: El índice de parentesco es calculado mediante la multiplicación de todos los índices de parentesco individuales de cada locus y dividiendo uno entre el valor obtenido de los índices de parentesco acumulado.
Al cotejar las muestras VB1 y C3 el índice de parentesco de todos los locus es 8.802144x10-6. El Índice de parentesco para estas dos muestras se obtuvo de:
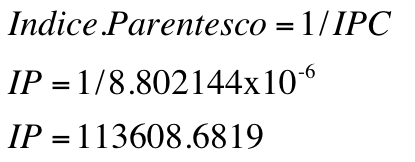

Resultando una probabilidad de parentesco entre estas dos muestras mayor al 99.9999% y el valor-p >0.0001 (valor de significancia), las muestras restantes fueron analizadas como la anterior y obteniendo los valores de igual manera, concentrando los resultados de los cálculos en la Tabla 8.
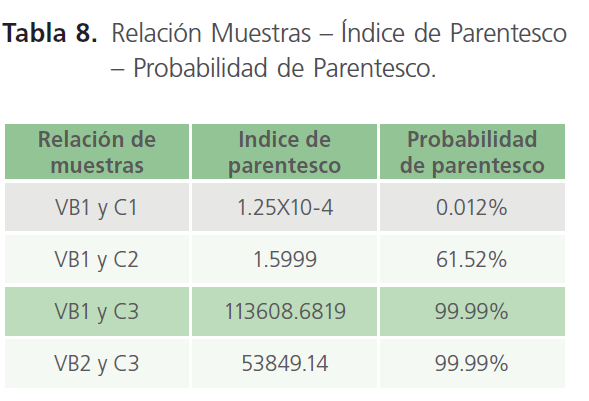
Tabla 8: Relación Muestras – Índice de Parentesco – Probabilidad de Parentesco.
Caso2. Índice de coincidencia
Problema: se presenta un caso donde el Problema de Estudio es Establecer el perfil genético y el índice de Coincidencia entre dos perfiles genéticos uno proveniente de un adolescente del sexo Masculino (XY) y el existente de las manchas de líquido hemático procedentes de unas prendas de vestir.
Material de estudio: se recibieron las muestras de referencia (Sangre en papel FTA® y cabello) del adolescente (Cabello) y las muestras biológicas procedentes de un arma punzo cortante tipo navaja (Arma), una playera blanca (PB), una camisa azul (CA) y una sudadera blanca (SB).
Análisis estadístico: Para realizas los cálculos estadísticos se empleó la Tabla 6 de frecuencias alélicas obtenida mediante el estudio de la Población Zacatecana. Los perfiles obtenidos y analizados se presentan en la Tabla 9 (anexa al final de la bibliografía) de manera simultánea se analizaron controles (+) y (-).
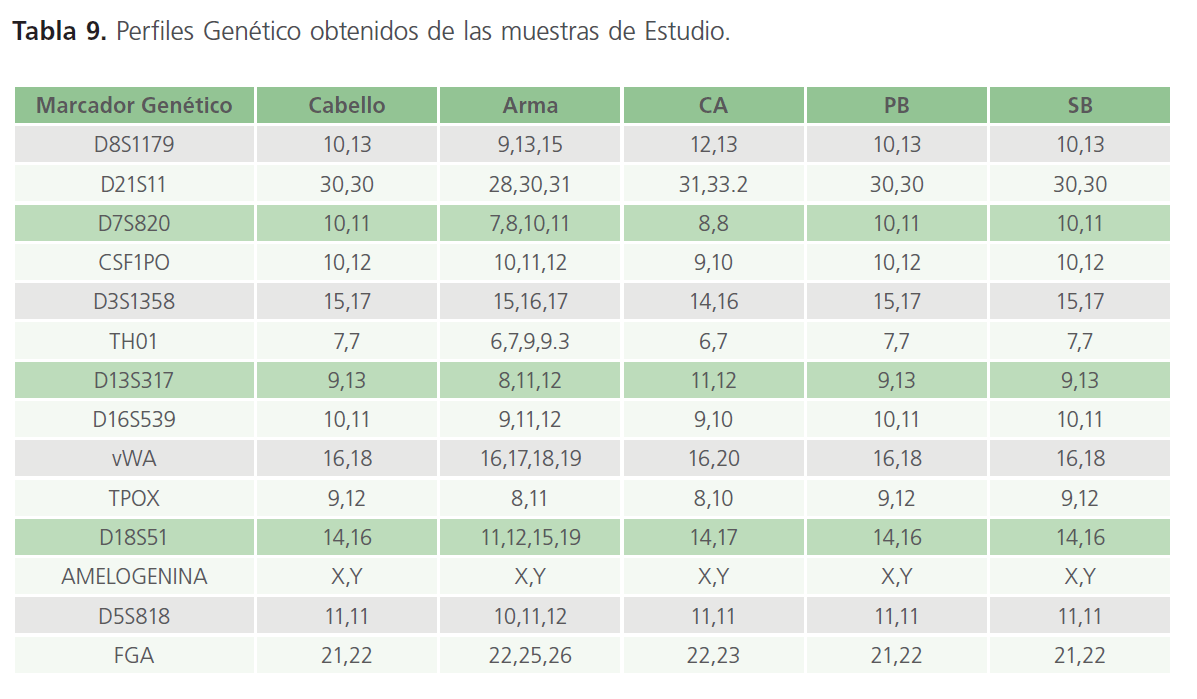
Tabla 9: Perfiles Genético obtenidos de las muestras de Estudio.
Posteriormente, se determina el Índice de Coincidencia entre el adolescente y las manchas rojas de líquido hemático levantadas de las prendas de vestir y el arma punzo cortante.
Resultados e interpretación: Los índices de coincidencia son calculados bajo la ley de equilibrio de Hardy-Weinberg evaluado como:
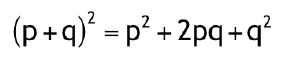
Donde p ≠ q
Aplicando las fórmulas anteriores para los perfiles obtenidos de las muestras analizadas se obtuvieron los datos contenidos en la Tabla 10.

Tabla 10: Relación Muestras – Índice de Coincidencia – Probabilidad de Coincidencia.
El índice de coincidencia es calculado mediante la multiplicación de los todos los índices de coincidencia individuales de cada locus y dividiendo uno entre el valor obtenido de los índices de coincidencia acumulado.
Al cotejar las muestras Cabello y PB el índice de coincidencia de todos los locus es 3.7048x10-11, el Índice de coincidencia para estas muestras se obtuvo a partir de:
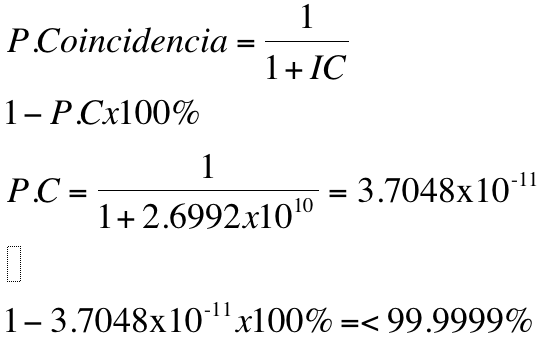

Debido a que los índices de coincidencia solo se calculan cuando son encontrados dos perfiles genéticos iguales, los índices de coincidencia resultan ser idénticos para las muestras obtenidas como: PB y SB comparadas con la muestra de referencia obtenida del adolescente obtenida de Cabello, con un índice de coincidencia entre estas muestras de 2.6999X10 [10] con una probabilidad de coincidencia mayor al 99.9999% entre estas muestras y un valor-p >0.0001, los valores obtenidos del análisis de estas muestra se encuentran concentrados en la Tabla 10.
Conclusiones
1. Se colectaron 271 muestras de los diferentes municipios del estado de Zacatecas en tarjetas FTA® (64.21%) e hisopados bucales (37.79%), de la muestras el 62.73% de ellas fueron obtenidas de personas del sexo masculino y el 37.27% restante del sexo femenino.
2. Tratando de cubrir todos los requisitos para la formulación de la base de datos y la extracción de ADN de las diferentes muestras usando la resina Chelex®100, la calidad y cantidad de ADN fue satisfactoria durante la amplificación. Debido a la sensibilidad de la extracción de ADN con la tarjeta FTA®, no es necesario realizar cuantificación de ADN ahorrando tiempo y costos para la obtención de perfiles genéticos.
3. Se evaluaron las frecuencias alélicas obtenidas con esta base de datos comparando con otros estudios como: las frecuencias alélicas de la población mestiza del noroeste de México, la base de datos de los hispanos contenida en el manual de Identifiler® con la que trabajan actualmente los laboratorios de Genética Forense cuyas diferencias pueden deberse: al número de individuos sometidos a estudio, mestizaje de población y a la información genética de generaciones anteriores.
4. Con este estudio podemos asegurar que las frecuencias alélicas obtenidas son exclusivamente de la población Zacatecana siguiendo las recomendaciones bibliografías, para la creación de bases de datos en las que describen que para la obtención de datos confiables, los individuos que participen en ello deberán preceder de al menos dos generaciones anteriores originarias del lugar de interés para el estudio. Característica que se siguió de manera estricta para el cumplimiento de datos exactos, rechazándose aquellas muestras cuyos requisitos no se cumplían en su totalidad. 5. Se logró obtener la base de datos de la Población Zacatecana, así como las frecuencias alélicas y genotípicas de la población, sumándose también otros parámetros de interés como: PM (permite saber que tan probable es encontrar dos genotipos que tomados al azar sean iguales), PD (indica la probabilidad de que dos individuos no relacionados y tomados al azar puedan ser diferenciados genéticamente), PIC (proporciona información sobre la heterocigosidad de cada marcador de acuerdo a la información obtenida de las frecuencias), TIP (el índice de paternidad nos indica la probabilidad que tiene el presunto padre de ser el padre biológico del hijo con respecto a una hombre tomado al azar), H(obs), H(esp) (las heterocigosidades muestran el porcentaje de heterocigotos que conforman la población y los datos esperados en el estudio haciéndose necesarios para evaluar el equilibrio de Hardy-Weinberg), H-W (el equilibrio de Hardy-Weinberg se analizó para observar el comportamiento de la población en estudio y saber si se encuentra en equilibrio o puede estar siendo afectada por algún factor como migración, mutación, selección natural ó deriva genética), X2 (permite calibrar las diferencias entre el número de individuos observados y los esperados contrastando dos hipótesis mutuamente excluyentes) realizando estos otros parámetros para complementar el análisis de este estudio.
6. Al llevar a cabo el análisis de la cantidad de Homocigotismo esperada y observada para calcular el equilibrio de Hardy-Weinberg se obtuvo que en 11 de los 15 marcadores genéticos como fueron: CSF1P0, D2S1338, D3S1358, D13S317, D16S539, TH01, TPOX, D18S51, D19S433, vWA, D21S11, el valor encontrado fue positivo indicándonos que existe un exceso de heterocigotos para los diferentes marcadores. Aplicando el Error Estándar (SE) para los datos obtenidos como cantidad de Heterocigotos Esperados para los diferentes marcadores la cantidad de marcadores con exceso de heterocigotos se redujo a un total de 8 marcadores genéticos.
7. Al observar los datos anteriores se calculó el Test de Chi-Cuadrada ó X2 que nos permite realizar una calibración de las diferencias entre el número de individuos esperados y observados y contrastar dos hipótesis las cuales son:
H0: la población del estado de Zacatecas se encuentra en equilibrio H-W.
H1: la población del estado de Zacatecas no está en equilibrio H-W.
Una vez observados los datos calculados para Chi- Cuadrada y analizando los datos para los grados de libertad en cada marcador analizado como en el caso de CSF1P0 donde X2 es igual a 2.55 y los grados de libertad para este marcador es 6. El dato para X2 es 12.59 con un valor crítico del 5 %, debido a que el valor crítico es mayor que el de Chi- Cuadrada nos hace concluir que no hay evidencia suficiente para rechazar la hipótesis H0 de que la población de Zacatecas esta en equilibrio de Hardy- Weinberg para el marcador CSF1P0. Aplicando estos criterios se analizaron de igual manera todos los marcadores restantes llegando a determinar que la población del estado de Zacatecas no se encuentra en equilibrio para dos de los marcadores analizados siendo estos: D21338 y D16S539. Encontrándose en equilibrio para los 13 marcadores restantes, obteniendo de manera exitosa la base de datos de la Población Zacatecana.
8. Al analizar las muestras obtenidas para el Caso 1 sobre el posible Vínculo Biológico entre las muestras obtenidas de los Vínculos Biológico 1(uno) y 2(dos) y las obtenidas de los cadáveres 1(uno), 2(dos) y 3(tres) se obtuvo el perfil genético y mediante el uso de la base de datos se logró obtener los índices de parentesco y la probabilidad de parentesco entre las muestras, para determinar la existencia de Parentesco entre las muestras VB1 y C3, así como también entre las muestras VB2 y C3 con un índice de parentesco de 99.999% y el valor-p >0.0001 (valor de significancia). Estos valores estadísticos indican que entre estas muestras SI EXISTE VÍNCULO BIOLÓGICO.
9. Al analizar las muestras obtenidas para el Caso 2 sobre el Índice de Coincidencia entre las muestras obtenidas del adolescente, las muestras obtenidas de las prendas de vestir y del objeto punzo cortante se obtuvo el perfil genético y mediante el uso de la base de datos se logró establecer los índices de coincidencia y la probabilidad de coincidencia entre las muestras, para determinar el índice de coincidencia entre las muestras Cabello, PB y SB el perfil genético que se encontró es el mismo con un índice de coincidencia de 2.6999x10 [10] entre estas, con una probabilidad de coincidencia mayor al 99.9999% y p>0.0001 (valor de significancia). Estos valores nos indican que estas muestras SON PROCEDENTES DE LA MISMA PERSONA.
11. El uso de la base de datos elaborada se empleó de manera exitosa brindándonos unos excelentes resultados para la aplicación en la práctica forense.
Agradecimientos
Este trabajo fue realizado en el Laboratorio de Genética Forense de la Procuraduría General de Justicia del Estado de Zacatecas y apoyado en su conjunto con la Procuraduría General de la República.
501
References
- Aranguren-Méndez, J.A., Román-Bravo, R., Isea, W., Villasmil, Y., Jordana, J. Los microsatélites (STR’s), marcadores moleculares de ADN por excelencia para programas de conservación: una revisión. Arch. Latinoam. Prod. Anim. 2005; 1 (13): 30-42.
- Biosystem Applied Manual de Usuario [Protocolo de usuario]. Genetic Analyzers 3130/3130xl.
- Butler, J.M. Forensic DNA Typing /Biology, Technology, and Genetics of STR markers [Sección del libro] = Combined DNA Index System // Forensic DNA Typing /Biology, Technology, and Genetics of STR markers. Elselvier, 2005.
- Butler, J.M. Forensic DNA Typing/Biology, Technology, and Genetics of STR markers [Sección del libro] // Forensic DNA Typing/Biology, Technology, and Genetics of STR markers. Elselvier, 2005.
- Butler, J.M. Forensic DNA Typing/Biology, Technology, and Genetics of STR markers [Sección del libro] = The Polymerase Chain Reaction (DNA Amplification) // Forensic DNA Typing / Biology, Technology, and Genetics of STR markers. Elselvier, 2005.
- Butler, J.M. DNA Biology Review, in forensic DNA Typing/ Biology, Technology, and Genetics of STR markers [Sección del libro] // Forensic DNA typing / aut. libro Butler John M. ed. USA. Elselvier, 2005.
- Cano Fernandez, J.A., Arce, A.B. https://www.iuisi.es/15_ boletines/15_2006/doc051-2006.pdf [En línea] = Genética Forense: Crimen e Identidad // https://www.iuisi.es/15_ boletines/15_2006/doc051-2006.pdf. [Consultado 9 de Octubre de 2012]. https://www.iuisi.es/15_boletines/15_2006/ doc051-2006.pdf. Instituto Universitario de Investigación sobre Seguridad Interior.
- Entrala, C. Técnicas de Análisis del ADN en Genética Forense [En línea] = Laboratorio de ADN forense, Depto. de Medicina Legal, Universidad de Granada España // Técnicas de Análisis del ADN en Genética Forense. - 2000. [Consultado 9 de Octubre de 2012]. https://www.ugr.es/~eianez/Biotecnologia/forensetec. htm.
- Gerard, J., Reynolds Grabowski, S. Principios de Anatomía y Fisiología [Sección del libro] = Aparato Reproductor // Principios de Anatomía y Fisiología / ed. González Jorge Alberto Ruíz. México: Oxford, 2002.
- González Andrade, F. Análisis Molecular de Variación de Polimorfismos STR autosómicos y de cromosoma “Y” en grupos étnicos de Ecuador con aplicación Médico Forense. = Análisis Molecular de Variación de Polimorfismos STR autosómicos y de cromosoma “Y” en grupos étnicos de Ecuador con aplicación Médico Forense. Zaragoza: [s.n.], 2006.
- Guradado Estrada, M. et al. Diversidad genética en la población mexicana: utilizacioón de marcadores de ADN. [Publicación periódica]. Mediagraphic ARtemisa. 2008; 3 (71): 162-174.
- McClean, Ph. Mendelian Genetics [En línea] = Mendelian Genetics // Mendelian Genetics. [Consultado 29 de septiembre de 2009]. https://www.ndsu.edu/pubweb/~mcclean/plsc431/ mendel/mendel4.htm.
- Norah Rudin, K.I. The Scientific Basis of DNA Typing [Sección del libro] = The Scientific Basis of DNA Typing // An introduccion to forensic DNA analysis / aut. libro Norah Rudin K. I. United States od America: McGraw-Hill, 2002.
- Novagen. I.a.a.o. M.K Proteinasa K [Protocolo de uso] = Proteinasa K // Proteinasa K Novagen. Darmstadt [s.n.], 2005.
- Promega. Promega Proteinasa K [Protocolo de Uso] // Proteinasa K Promega. 2008.
- Quintero Ramos, A. et al. Uso de marcadores polimoroficos = Uso de marcadores polomórficos. 2005.
- Rodriguez Carlin, C. et al. Genética Forense [Publicación periódica] // Fuente. – México, 2010.
- Watson, J.D. et al. Biología Molecular del Gen [Publicación periódica] = Técnicas de Biología Molecular. Panamericana, 2005.
- Wing Kam, F., Yue-qing, H. Statistical DNA Fonesics, Theory, Methods and Computation Statistics in Practice [Sección del libro] // Statistical DNA Fonesics, Theory, Methods and Computation Statistics in Practice. Ed. Wiley. 2008 .