Keywords
Blue crab, Callinectes sapidus, Cytochrome oxidase I, Mitochondrial DNA, Molecular phylogeny, Population genetics
Özet
Türkiye’nin Akdeniz kıyılarında bulunan mavi yengeç (Callinectes sapidus) populasyonları arasındaki genetik çeşitliliğin ortaya çıkarılmasında mitokondriyal sitokrom c oksidaz I (COI) geninin nükleotid dizisi farklılıklarından yararlanılmıştır. Akdeniz kıyısı boyunca, 8 farklı po-pulasyondan (Akyatan, Fethiye, Beymelek, Güllük, İskenderun, Köyceğiz, Mersin ve Taşucu) elde edilen 143 örnek nükleotid kompozisyonu ve nükleotid çifti frekansı bakımından incelen-miştir. DNA dizilerinin farklılığı kullanılarak tespit edilen genetik farklılığın belirlenmesinde Tamura 3-parameter modeli kullanılmıştır. DNA dizi analizi gerçekleştirilen 143 örnek göz önüne alındığında, 655 baz çitlik bölgede 42 (%6.41) değişken bölge ve ortalama %1.9 varyas-yon saptanmıştır. En yüksek genetik farlılık %2.5 ile Akyatan-Güllük populasyonları arasında tespit edilmiş ve ortalama R değeri tüm populasyonlar için 3.59 olarak hesaplanmıştır. Filoge-netik ağacın oluşturulmasında unweighted pair-group method of arithmetic average (UPGMA) yöntemi kullanılmıştır. Dal uzunlukları toplamı= 0.07232 olarak hesaplanan filogenetik ağaç değerlendirildiğinde, mavi yengeç populasyonları arasında coğrafik uzaklıkla korele bir genetik gruplaşma olduğu sonucuna varılmıştır.
Anahtar Kelimeler
Callinectes sapidus, Mavi yengeç, Mitokondriyal DNA, Moleküler filogenetik, Populasyon genetiği, Sitokrom oksidaz I.
Giriş
Mavi yengeç (Callinectes sapidus), Atlantik Okyanusu’nun batısı boyunca bulunan nehir ağızları ve deniz ortamlarında dağılım gösteren, Decapoda takımından, ekolojik ve ekonomik öneme sahip bir türdür. Dağılımları Nova Sco-tia’dan Arjantin’in kuzeyine kadar uzanmakta, daha kuzey ve güneyinde ise mevsimsel olarak değişiklik göstermektedir (Williams 1984). Ja-ponya ve Avrupa sularına gemilerin balast suları ile giriş yapan bu tür, CIESM raporlarına göre Baltık Denizi’nde, Kuzey Denizi’nde, Karade-niz’de ve Akdeniz’de yerleşik populasyonlar oluşturmuştur.
Mavi yengeçlerin jenerasyon süresi yaklaşık olarak bir yıldır (Williams 1984). En belirgin po-pulasyon kayıpları ilk yıl içerisinde gözlemlenir ve yaşam süreleri en fazla 6 yıl olarak belirlen-miştir (Pellegrini et al. 2001). Türün yayılımı ge-nel olarak yaşam evresine ve coğrafik konumuna göre değişkenlik göstermektedir. Larval gelişi-mini kıyı bölgelerinde geçiren mavi yengeçlerin olası yayılımları üzerinde larval davranışlarının ve mevsimsel rüzgarların etkisinin büyük olduğu bilinmektedir (Epifanio and Garvine 2001). Do-ğal olarak bulundukları Kuzey Amerika kıyıla-rındaki yetişkin mavi yengeçlerin dağılım alanı genel olarak birkaç nehir ağızı ve etraftaki kıyısal bölgelerle kısıtlı kalmaktadır (Benefield ve Lin-ton 1990).
Ekolojik ve ekonomik olarak büyük öneme sahip olan mavi yengeçlerin populasyon yapıları-nın genetik analizine yönelik çalışmaların, şaşır-tıcı bir şekilde fazla üzerinde durulmamış ve bu konuda, doğal populasyonlar dışında çok fazla çalışma yapılmamıştır. Oysa moleküler teknikle-rin deniz canlılarının populasyon yapılarının ge-netik karakterizasyonunda başarıyla uygulandığı bilinmekte, fakat Decapoda takımından karides-ler, ıstakozlar ve yengeçler üzerinde yoğunlaşan çalışmalara çok fazla rastlanmamaktadır.
Mitokondriyal DNA, rekombinasyon olma-ması, maternal kalıtım özelliği ve nükleer ge-noma oranla daha hızlı evrimsel değişim oranın-dan dolayı, dekapodların filogenetik ilişkilerin ortaya koyulmasında ve tür tanımlanmasında en çok kullanılan moleküler belirteç olarak karşı-mıza çıkmaktadır (McMillen-Jackson and Bert 2004). Ayrıca bu belirteçler deniz canlılarının larval dağılımını ve gen akışını dolaylı olarak ortaya koymada başarılı sonuçlar vermiştir (Hamm and Burton 2000). Deniz canlılarında, mitokondriyal genomlar genellikle kompakt ya-pıda (15-20 kb), aynı gen sıralamasına sahip ve replikasyon ile transkripsiyonu kontrol eden kodlama yapmayan bir bölgeden oluşan dairesel bir DNA zincirinden meydana gelmektedir (Bo-ore 1999). Birçok türün mitokondriyal DNA’sı, nükleotid değişiklikleri veya kısa dizi değişiklik-leri içermesine rağmen gen dizilimleri uzun sü-reler boyunca stabil kalmıştır (Boore and Brown 1995).
Mitokondriyal genlerden sitokrom c oksidaz I (COI), tür ayrımında ve populasyon yapısının belirlenmesinde en çok kullanılan moleküler be-lirteçlerden biri olarak karşımıza çıkmaktadır. Bu genin yaklaşık 655 baz çiftlik bölgesi “DNA bar-kodu” olarak da bilinmektedir. DNA barkodlama son yıllarda birçok genetik çalışmada karşımıza çıkmaktadır (Undheim et al. 2010). Bu çalışmada amacımız, ilk olarak Türkiye’nin Akdeniz kıyı-ları boyunca dağılım gösteren mavi yengeç po-pulasyonları arasında genetik bir farklılık olup olmadığının ortaya çıkarılması ve ardından bunun tespit edilmesinde COI gen dizilerinin analizinin uygun bir moleküler belirteç olup olmadığının belirlenmesidir.
Materyal ve Metot
Çalışmada, Akdeniz kıyısı boyunca belirlenen 8 istasyondan toplam 143 örnek elde edilmiştir. Örnekler buz üzerinde laboratuvara getirilmiş, kalp ve kas dokusu örnekleri alındıktan sonra DNA izolasyonları gerçekleştirilene kadar %95 etanol içerisinde -20°C’de saklanmıştır.
Doku örnekleri sıvı nitrojen ile dondurularak, porselen havan yardımıyla toz haline getirilmiş-tir. Toz haline getirilen dokulardan DNA izolas-yonu Quiagen DNeasy Blood & Tissue Kit ile üreticinin protokolüne uygun olarak gerçekleşti-rilmiştir. Elde edilen DNA’ların kalite ve mikta-rını saptamak amacıyla NanoDrop ND – 1000 spektrofotometre'den yararlanılmıştır. Kalıp ola-rak kullanılacak DNA’lar bekletmeden polimeraz zincir reaksiyonunda (PCR) kullanılmıştır.
Mitokondriyal COI geninin yaklaşık 700 baz çiftlik bölümünün PCR ile çoğaltılmasında LCO1490f (5’-GGTCAACAAATCATAAAGATATTGG-3’) ve HCO2198r (5’-TAAACTTCAGGGTGACCAAAAAATCA-3’) primer çiftinden yararlanılmıştır (Folmer et al. 1994). PCR reaksiyonları, son hacmi 50 μl olan reaksiyonlar halinde ve içeriği: 5 μl 10X Taq Buffer +KCl (100 mM Tris-HCl, 500 mM KCl, pH 8.8), 4μl MgCl2 (25mM), 1.25 μl dNTP (10 mM), her primerden 2 μl (10 pM/μl), 2.5 U Taq polimeraz (5U/μl) ve 2 μl DNA (50 ng/μl) olacak şekilde ayarlanmıştır. PCR reaksiyonları MJ Re-search DNA Engine Tetrad2 termal döngü ciha-zında, negatif kontrol kullanılarak 95°C’de 2 da-kikalık bir ön denatürasyon aşamasını takiben 35 döngülük 95°C’de 1 dakika, 50°C’de 1 dakika, 72°C’de 1 dakika ve son olarak 72’de 10 dakika-lık program kullanılarak gerçekleştirilmiştir. Elde edilen PCR ürünleri %2’lik agaroz jelde yürütül-dükten sonra UV görüntüleyici altında incelene-rek bant boyları kontrol edilmiştir. PCR ürünleri-nin DNA dizi analizi reaksiyonları öncesinde pü-rifikasyonunda Wizard SV Gel and PCR Clean- Up System, üreticinin sağladığı protokol doğrul-tusunda kullanılmıştır. Temizlenmiş PCR ürünle-rinin dizi analizleri Beckman Coulter CEQ 8000 cihazında, Beckman Coulter CEQ Dye Termina-tor Cycle Sequencing Kit kullanılarak gerçekleş-tirilmiştir.
Dizi analizi sonrasında elde edilen nükleotid dizileri GenBank’dan elde edilen referans diziler kullanılarak Sequencher 5.0 programında hiza-lanmış, okuma hatası nedeniyle oluşabilecek in-sersiyon ve delesyonlar kontrol edilmiştir. Diziler hizalandıktan sonra GenBank veri tabanına kayıt ettirilmiştir (JN561325-JN561332). Her örnek için nükleotid kompozisyonu, korunmuş bölge, değişken bölge, tekrar bölgeleri ile parsimoni anlamlı bölgelerin analizi gerçekleştirilmiş ve nükleotid çifti frekansları hesaplanmıştır. Subs-titüsyon ile transisyon/transversiyon oranının (R) hesaplanmasında Tamura 3-parameter modeli kullanılmıştır. Pairwise genetik uzaklık matrisi oluşturulmuş ve populasyonlar arasındaki genetik uzaklık diziler arasındaki baz substitüsyonları göz önünde bulundurularak Tamura 3-parameter modeline uygun olarak hesaplanmıştır.
Filogenetik ağacın oluşturulmasında unwe-ighted pair – group method of arithmetic average (UPGMA) yöntemi kullanılmıştır. Ağacın oluştu-rulmasında kullanılan evrimsel uzaklıkların he-saplanması Tamura 3-parameter modeline göre gerçekleştirilmiştir. Elde edilen filogenetik ağa-cın güvenilirliğinin tespitinde 1000 tekrarlı bo-otstrap testlerinden yararlanılmıştır. Tüm filoge-netik analizler MEGA 5 programında gerçekleşti-rilmiştir.
Bulgular ve Tartışma
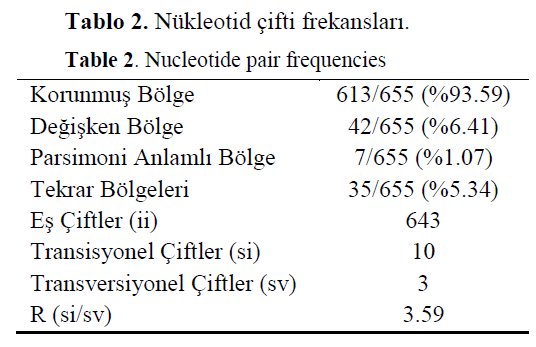
Toplam 143 örneğe ait COI gen dizisi analizi gerçekleştirilmiş ve 8 populasyon arasındaki ge-netik farklılık ortaya koyulmuştur. Aynı populas-yona ait bireylerin dizi analizi sonuçları birebir örtüştüğünden her populasyon tek bir örneğin COI dizisi ile temsil edilmiştir. G-C oranlarına bakıldığında (Tablo 1) değerlerin %38.8-39.8 arasında değişkenlik gösterdiği saptanmış ve or-talama G-C oranı %39.3 olarak belirlenmiştir. Tüm örnekler göz önüne alındığında 655 baz çiftlik bölgede 42 nükleotidde değişiklik olduğu (%6.41) görülmüştür. Transisyon/transversiyon oranı (R), eş çiftler, tekrar bölgeleri ve parsimoni anlamlı bölgeler Tablo 2’de verilmiştir.
Tablo 1. Nükleotid kompozisyonu (%).
Table 1. Nucleotide composition (%)
Tablo 2. Nükleotid çifti frekansları.
Table 2. Nucleotide pair frequencies
Substitüsyon oranlarının maximum likelihood yöntemiyle belirlenmesinde Tamura 3-parameter modeli kullanılmıştır (Tablo 3). Substitüsyon oranları bir bazdan diğerine substitüsyon geçiş ihtimalini temsil etmektedir. Transisyonel subs-titüsyon oranları A/G ve T/C için 15.34, C/T ve G/A için ise 23.71 olarak hesaplanmıştır. Trans-versiyonel oranlar Tablo 3’te verilmiştir. Bu so-nuçlar ışığında hesaplanan transis-yon/transversiyon oranı (R) 3.40 olarak hesap-lanmıştır. Hesaplamaların yapılmasında kullanı-lan nükleotid frekansları A ve T için %30.35, G ve C için %19.65’dir.
Tablo 3. Substitüsyon matrisi*.
Table 3. Nucleotide substitution matrix
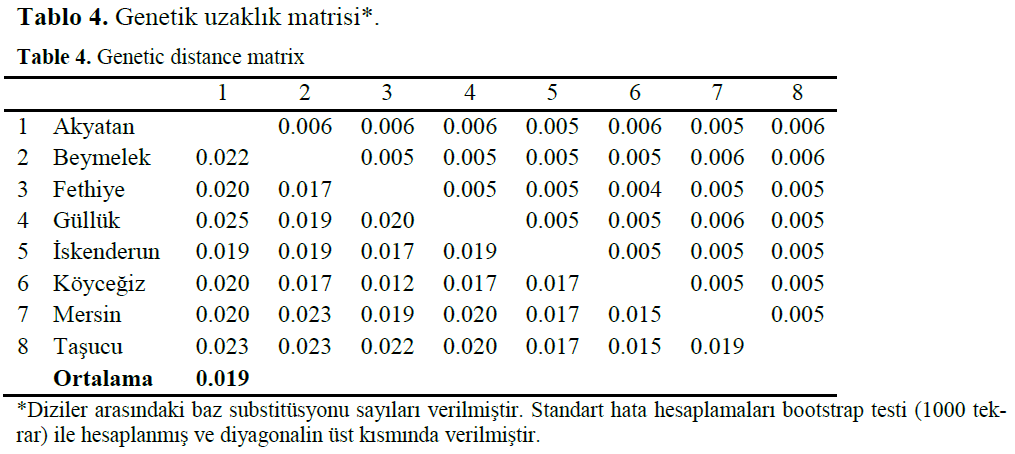
Pairwise genetik uzaklık matrisi Tamura 3-pa-rameter modeline göre oluşturulmuştur (Tablo 4). Populasyonlar arasındaki evrimsel değişimin he-saplanmasında diziler arasındaki baz substitüsyon sayıları kullanılmıştır. Mavi yengeç populasyon-ları arasındaki ortalama genetik uzaklık 0.019 (1.9%) olarak bulunmuştur. Elde edilen sonuçlar populasyonlar arasındaki genetik farklılığın po-pulasyonların bulunduğu coğrafik alanların uzaklığı ile korele olarak artış gösterdiğini işaret etmektedir. En yüksek nükleotid farklılığı Güllük ve Akyatan populasyonları arasında 0.025 (%2.5) olarak saptanmıştır. En düşük genetik farklılık ise birbirine daha yakın populasyonlar olan Köyce-ğiz ve Fethiye populasyonları arasında 0.012 (%1.2) olarak hesaplanmıştır. Diğer populasyon-lardan en belirgin biçimde ayrılan mavi yengeç populasyonu ise 0.021 (%2.1) genetik uzaklık değeri ile Akyatan populasyonu olarak belirlen-miştir. Genetik uzaklık sonuçları 143 mavi yen-geç örneğinin dizilerinin pairwise analizlerine göre hesaplanmış ve standart hataların hesaplan-masında 1000 tekrarlı bootstrap testlerinden fay-dalanılmıştır (Tablo 4).
Tablo 4. Genetik uzaklık matrisi*.
Table 4. Genetic distance matrix
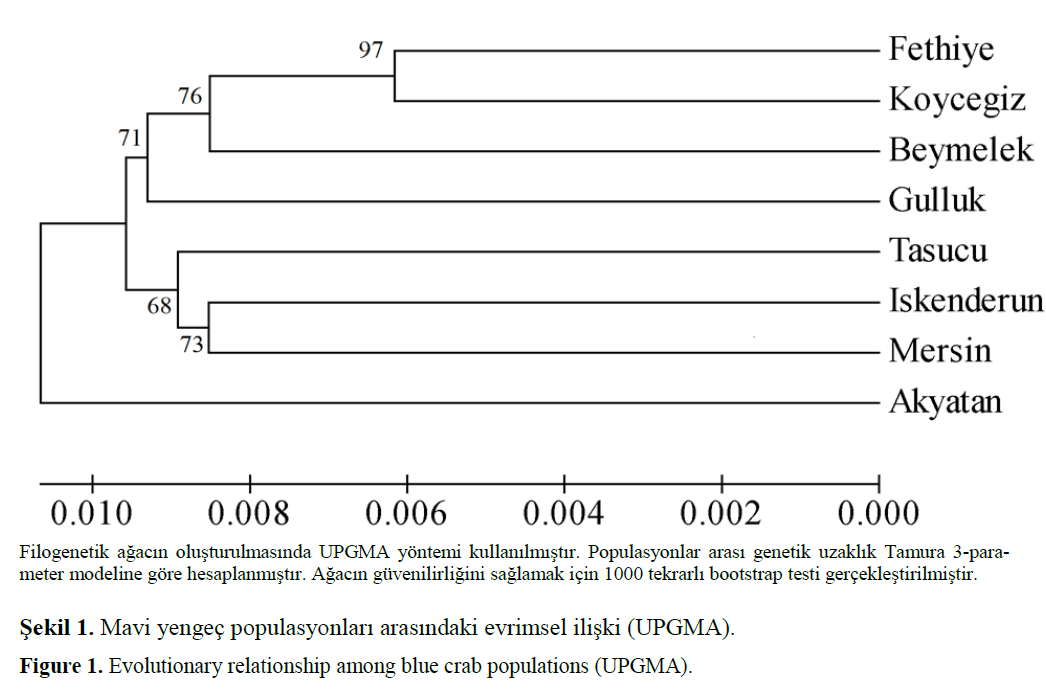
Mavi yengeç populasyonlarının evrimsel iliş-kilerinin hesaplanmasında ve filogenetik ağacın oluşturulmasında UPGMA yöntemi kullanılmış-tır. Populasyonlar arası genetik uzaklıkların be-lirlenmesinde ise Tamura 3-parameter yöntemin-den yararlanılmıştır. Ağacın güvenilirliğini sağ-lamak için 1000 tekrarlı bootstrap testi gerçek-leştirilmiştir. Evrimsel geçmiş analizlerinde tüm örneklerin nükleotid veri setlerinden yararlanıl-mıştır. Toplam dal uzunluğu 0.07232 olarak he-saplanan filogenetik ağaç Şekil 1’de gösterilmiş-tir.
Şekil 1. Mavi yengeç populasyonları arasındaki evrimsel ilişki (UPGMA).
Figure 1. Evolutionary relationship among blue crab populations (UPGMA).
Akdeniz boyunca farklı alanlarda populas-yonlar oluşturan mavi yengeçlerin mitokondriyal COI dizilerinden elde edilen sonuçlar, türün Ak-deniz’e yerleşmesinin ardından farklı coğrafik konumlarda genetik farklılıkları bulunan popu-lasyonlar oluşturduğunu göstermektedir. Bu ça-lışmada belirteç olarak kullanılan COI geni, sık-lıkla kullanılan 12S ve 16S rRNA’ya kıyasla ko-donlarındaki üçüncü nükleotidin daha yüksek oranda substitüsyon göstermesinden dolayı 3 kat daha yüksek bir evrim hızına sahip olması (Knowlton and Weigt 1998) nedeni ile tercih edilmiştir. Bu evrim hızı, COI dizilerinin sadece birbirine yakın türlerin ayrımında değil, aynı za-manda bir türün farklı populasyonlarının ayrı-mında da başarılı bir moleküler belirteç olarak kullanılmasını sağlamaktadır (Cox and Hebert 2001).
Çalışmadan elde edilen sonuçlar, mitokondri-yal COI geninin amino asit dizilerinin mavi yen-geç populasyonları arasındaki filogenetik sinyali tespit etmede başarılı olduğunu göstermektedir. Mavi yengeç örneklerinin ortalama nükleotid kompozisyonlarından da görüldüğü üzere (Tablo 1) COI geni yüksek A-T oranı bakımından bir seleksiyonun etkisi altında bulunmaktadır. Bu bölgedeki yüksek A-T içeriği COI genini popu-lasyon seviyesinde bile hassas bir moleküler be-lirteç yapmaktadır (Sanchis et al. 2001). Bu ça-lışmada, mavi yengeç populasyonlarının ortalama A-T oranı %60.7 olarak tespit edilmiştir. Örnek-leme yapılan 8 populasyonun A-T değerlerinin farklı olması ise geçmişte meydana gelmiş olası coğrafik izolasyonlar ile açıklanabilmektedir.
COI geninin 655 baz çiftlik bu bölgesinin mavi yengeç populasyonlarının filogenetik fark-lılıklarını tespit etmede başarılı bir moleküler be-lirteç olduğunun bir başka göstergesi de nükleo-tid çifti frekansı analizi sonrasında tespit edilen %6.41’lik değişken bölge oranıdır (Tablo 2). Substitüsyon oranları incelendiğinde, en yüksek oranın C/T ve G/A değişimlerinde tespit edilen %23.71 olduğunu görmekteyiz. C-G den A-T’ye bu yüksek oranlı geçişlerin sonuçları, nükleotid kompozisyonu analizinde gördüğümüz yüksek A-T oranını açıklamaktadır. Substitüsyon matrisinin Tamura 3-parameter modeli kullanılarak yapılan tahmininde elde edilen transisyon/transversiyon oranının (R) 3.40 ile nükleotid çifti frekansı son-rası elde edilen 3.59’luk R değerinden küçük ve ona yakın çıkması sonuçların güvenilirliği açısın-dan önemli bir detay olarak değerlendirilmiştir.
Türkiye’nin Akdeniz sahillerinden elde edilen mavi yengeç örnekleri arasında belirgin bir gene-tik uzaklık göze çarpmaktadır (Tablo 4). Mavi yengeç populasyonları arasındaki bu genetik uzaklık 0.012 ile 0.025 arasında, ortalama 0.019'luk bir değişkenlik göstermektedir. Genetik uzaklık bakımından birbirinden en çok ayrılan populasyonlar Akyatan-Güllük populasyonları olarak saptanırken (0.025), birbirine en yakın po-pulasyonlar Köyceğiz-Fethiye populasyonları (0.012) olarak tespit edilmiştir. Akyatan populas-yonu diğer tüm populasyonlara olan ortalama 0.021’lik genetik uzaklık değeri ile en farklı po-pulasyon olarak gözükmektedir.
Dal uzunluk toplamı 0.07232 olan ağaçtaki kümelenmeler dikkatli olarak incelendiğinde ge-netik uzaklık ile coğrafik uzaklık arasında korele bir ilişki göze çarpmaktadır (Şekil 1). Filogenetik ağaç incelendiğinde 2 ana dala ayrıldığı görül-mektedir. Birinci ana dalda genetik uzaklık ana-lizleriyle uyumlu bir şekilde Akyatan populasyo-nunun diğerlerinden ayrıldığı, diğer 7 populasyo-nun ikinci bir dalda kümelendiği görülmektedir. İkinci ana dal kendi içinde 2 dala ayrılmaktadır. Birinci dalda Akyatan populasyonuna coğrafik olarak daha yakın olan İskenderun, Mersin ve Taşucu populasyonları kümelenmiştir. Bunlar da kendi arasında İskenderun ve Mersin ayrı bir alt dal, Taşucu ayrı bir alt dal oluşturacak şekilde kümelenmiştir. İkinci dalda ise daha batı da bu-lunan diğer 4 populasyon kümelenmiştir. Burada dikkat çeken nokta ise Güllük ve Beymelek po-pulasyonlarının ayrı alt dallarda, Köyceğiz ve Fethiye populasyonları ise birbirinden belirgin biçimde ayrılmış olmakla beraber aynı alt dalda kümelenmiş olmasıdır. Dalların yanlarında bo-otstrap testleri (1000 tekrar) sonucunda oluşturu-lan tekrar ağaçlarının yüzde oranı verilmiştir (Şe-kil 1). Hesaplanan tüm bootstrap değerlerinin %68 seviyesinde ve üzerinde olduğu görülmekte ve %70 in üzerindeki bootstrap değerlerinin oluşturduğu gruplandırmanın %95’in üzerinde bir doğruluğa sahip olduğunu bilinmektedir (Hillis and Bull 1993). Filogenetik ağaçta sadece bir bo-otstrap değeri %70’den küçük (%68) olarak bu-lunmuştur.
Sonuç
Türkiye’nin Akdeniz kıyısı boyunca 8 farklı istasyondan örnekleme yapılan bu çalışmada, mavi yengeç populasyonlarının COI gen dizileri arasında belirgin farklılıklar tespit edilmiştir. Elde edilen sonuçlar hem mavi yengeç populas-yonlarının genetik olarak değerlendirilmesinde ilk kez COI geni kullanılması bakımından, hem de Akdeniz kıyısında yerleşik populasyonlar oluşturan mavi yengeçlerin moleküler ayrımına yönelik ilk çalışma olması bakımından önem ta-şımaktadır. Gerek populasyonlar arasındaki A-T değerleri arasındaki farklılık, gerekse genetik uzaklık değerlerinin ve bununla ilişkili olarak populasyonların filogenetik ağaçtaki yerleşimle-rinin coğrafik dağılımla uyumlu olması, mavi yengeçlerin uygun olmayan çevre koşullarında bulundukları yerden çok fazla dağılım gösterme-meleri sonucunda oluşan coğrafik izolasyonlar ile açıklanabilmektedir. Mavi yengeç populasyonla-rına yönelik tüm Akdeniz’den örnekleme yapıla-cak şekilde genişletilecek bir çalışma bu konuda daha detaylı sonuçlar elde etmemize yardımcı olacaktır.
Kaynaklar
Benefield, R.L., Linton, T., (1990). Movement study of blue crabs in Trinity Bay. Texas Parks and Wildlife Department, Coastal Fisheries Division, Management Data Se-ries, 16: 1-11.
Boore, J.L., (1999). Animal mitochondrial geno-mes, Nucleic Acids Research, 27: 1767-1780.doi: 10.1093/nar/27.8.1767
Boore, J.L., Brown, W.M., (1995). Complete sequence of the mitochondrial DNA of the annelid worm Lumbricusterrestris, Gene-tics, 141: 305-319.
Cox, A.J., Hebert, P.D.N., (2001). Colonization, extinction and phylogeographic patterning in a freshwater crustacean, Molecular Ecology, 10: 371-386.doi: 10.1046/j.1365-294x.2001.01188.x
Epifanio, C.E., Garvine, R.W., (2001). Larval transport on the Atlantic continental shelf of North America: a review, Estuarine, Coastal and Shelf Science, 52: 51-77.doi: 10.1006/ecss.2000.0727
Folmer, O., Black, M., Hoeh, W., Lutz, R., Vri-jenhoek, R., (1994). DNA primers for ampli-fication of mitochondrial cytochrome c oxidase subunit I from diverse metazoan in-vertebrates, Molecular Marine Biology and Biotechnology, 3: 294-299.
Hamm, D.E., Burton, R.S., (2000). Population genetics of black abalone, Haliotiscrachero-dii, along the central California coast, Jour-nal of Experimental Marine Biology and Ecology, 254: 235-247.doi: 10.1016/S0022-0981(00)00283-5
Hillis, D.M., Bull, J.J., (1993). An empirical test of bootstrapping as a method for assessing confidence on phylogenetic analysis, Syste-matic Biology, 42: 182-192.
Knowlton, N., Weigt, L.A., (1998). New dates and new rates for divergence across the Isthmus of Panama. Proceedings of the Ro-yal Society of London. Series B, 265: 2257-2263 doi: 10.1098/rspb.1998.0568
McMillen-Jackson, A.L., Bert, T.M., (2004). Mitochondrial DNA variation and popula-tion genetic structure of the blue crab Calli-nectes sapidus in the eastern United States, Marine Biology, 145: 769-777.
Pellegrin, G.Jr., Guillory, V., Prejean, P., Perry, H., Warren, J., Steele, P., Wagner, T., Heath, S., (2001). Length-based estimates of total mortality for Gulf of Mexico blue crab, Pro-ceedings of the Blue Crab Mortality Sympo-sium, 28–29 May 1999, Lafayette. Louisiana Gulf States Marine Fisheries Commission, 90: 42-49.
Sanchis, A., Michelena, J.M., Lattore, A., Quicke, D.L., Gardenfors, U., Belshaw, R., (2001). The phylogenetic analysis of vari-able-length sequence data: elongation factor-1 alpha introns in European populations of the parasitoid wasp genus Pauesia (Hyme-noptera: Braconidae: Aphidiinae), Molecu-lar Biology and Evolution, 18(6): 1117-1131.
doi:10.1093/oxfordjournals.molbev.a003882
Undheim, E.A.B., Norman J.A., Thoen, H.H., Fry, B.G., (2010). Genetic identification of Southern Ocean octopod samples using mtCOI, ComptesRendusBiologies, 333: 395-404.doi: 10.1016/j.crvi.2010.02.002
Williams, A.B., (1984). Shrimps, lobsters, and crabs of the Atlantic coast of the eastern United States, Maine to Florida. Smithso-nian Institution Press, Washington, D.C.652